宜居环境演化和生命信号探寻是火星探测的核心科学目标。通过实施原位探测和采样返回任务,可以直接获取火星原位科学数据和火星表面样品,寻找火星潜在生命信号。与此同时,地球上的一些极端环境因与火星条件高度相似(如极端干旱、高辐射、高盐碱等)被称为类火星环境,系统研究这些类火星极端环境中的微生物及其生存策略有助于评估火星过去或现今存在生命的可能性、遴选火星生命信号探测的优先区域、筛选生命探测的信号指标、优化生命信号探测的方法和策略(图1)。

图1 类比火星不同地质历史时期的地球典型类火星环境和火星的潜在宜居区域(刘立和林巍, 2025,第四纪研究)
柴达木盆地位于我国青藏高原北部,是全球海拔最高、最干旱的荒漠之一。恶劣的环境条件(极端干旱、盐碱化土壤、高紫外辐射、寒冷等)以及广泛分布的类火星地貌(冲沟、冲积扇、泥石流、雅丹、沙丘等)和蒸发盐矿物(硫酸盐、氯化物等),使得柴达木盆地成为一处典型且独特的类火星地区。微生物是柴达木盆地的主要生物类群,构建柴达木盆地微生物基因组资源数据库,将为研究类火星极端环境生态系统、挖掘耐极端环境菌株/基因资源、探究生命在极端环境下的适应机制等提供数据支持,也为我国火星原位探测和采样返回任务中的生命信号识别表征和污染防控等提供科学依据。
中国科学院地质与地球物理研究所行星科学与前沿技术重点实验室的刘立博士后与合作导师林巍研究员等,系统采集了柴达木盆地代表性类火星地貌和不同深度的土壤样品,利用宏基因组学技术,成功获得了1773条细菌和古菌基因组以及2060条病毒基因组,并构建了柴达木盆地类火星环境微生物基因组资源数据库(图2)。在此基础上,结合土壤理化性质、矿物组成等地质环境信息,系统研究了柴达木盆地不同地貌类型和土壤深度中微生物的多样性、分布特征、生存策略及其相互作用关系。

图2 柴达木盆地微生物基因组数据库。a.柴达木盆地采样地点类型与位置;b.1773条细菌和古菌基因组质量情况;c.2060条病毒基因组质量情况
柴达木盆地丰度最高的细菌、古菌和病毒类群分别属于放线菌门(Actinobacteriota)、嗜盐古菌门(Halobacteriota)和尾噬菌体门(Uroviricota)(图3a)。超过94%的基因组属于先前未被分类鉴定的新物种,表明柴达木盆地类火星极端环境中存在着大量未知的微生物“暗物质”。相关性分析结果显示,土壤水分、pH值和矿物组成等显著影响微生物的群落结构和空间分布(图3b)。其中,柴达木土壤中病毒群落以裂解型病毒为主,且具有跨宿主侵染特征(即一种病毒可侵染多种宿主)(图4a、图4b)。在生物量稀少的柴达木盆地,这种跨宿主侵染特征使病毒在类火星极端环境中具有更强的适应性。病毒宿主预测结果发现,柴达木盆地的病毒主要侵染优势微生物类群(图4a),通过“Kill-The-Winner”机制(即病毒优先裂解环境中的高丰度宿主)来调控微生物多样性和群落稳定性。

图3 柴达木盆地原核生物(细菌和古菌)和病毒群落。a.原核生物和病毒群落组成;b.原核生物和病毒群落结构和功能与各环境因子的相关性分析
超过38%的细菌和古菌基因组具有CO氧化的关键基因coxSML,10%以上具有催化H2氧化的[NiFe]-/[FeFe]-氢化酶基因,表明在营养匮乏的柴达木盆地土壤中,细菌和古菌可以通过利用大气中的痕量气体获取能量。有超过一半的细菌和古菌被鉴定为病毒的宿主,且具有复杂的代谢潜力,表明病毒可能通过侵染这些宿主种群,从而参与到柴达木盆地的生物地球化学循环中。此外,研究团队鉴定出超过32000个潜在的病毒辅助代谢基因(AMGs),涵盖碳循环、光合作用和磷循环等关键代谢通路,揭示出病毒可能通过AMGs调控宿主代谢并驱动生物地球化学过程(图4c)。还发现了涉及合成抗生素、色素和生物碱的AMGs,病毒可能通过这些AMGs增强宿主在类火星极端环境中的生存能力,通过提高宿主的环境适应性从而增强病毒自身的存活概率。
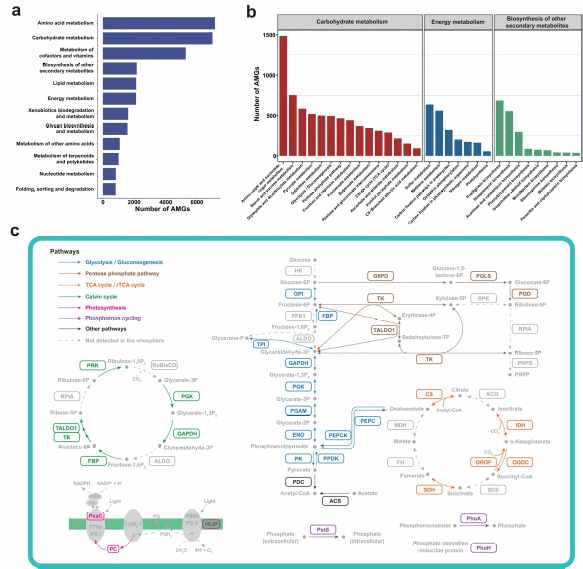
图4 病毒宿主预测和辅助代谢基因鉴定。a.病毒-宿主关系预测结果;b.广宿主病毒筛选;c.涉及生物地球化学循环通路的病毒辅助代谢基因鉴定
该研究构建了柴达木盆地微生物的基因组资源数据库,揭示了类火星极端环境中微生物的多样性、地理分布与功能潜力,发现病毒在提升宿主的极端环境适应性和驱动生物地球化学循环中具有重要作用,为类火星环境的天体生物学研究提供了数据支撑。
研究成果分别发表于国际学术期刊Scientific Data和Environmental Microbiology。研究受国家自然科学基金资助。
1. 刘立,王喆,张文斯,林巍*. Recovery of 1,773 microbial genomes and 2,060 viral genomes from the Mars-analog Qaidam Basin desert[J]. Scientific Data. 2025, 12: 1795. DOI: 10.1038/s41597-025-06085-3.
2. 刘立,王喆,张文斯,周之超,林巍*. The hidden viral network: How viruses shape microbial adaptability and biogeochemical cycling in the polyextreme Mars-analog Qaidam Basin desert[J]. Environmental Microbiology. 2025, 27: e70202. DOI: 10.1111/1462-2920.70202.

刘立(博士后)

